[서울=뉴스핌] 김영섭 기자 = 과학기술정보통신부는 연세대 의과대학 김상우 교수 연구팀이 환자의 암세포 시료를 분석할 때 외부요인을 줄여 분석의 정확도를 높이는 방법을 개발했다고 11일 밝혔다.
연구결과는 유전체학 분야 국제학술지 '지놈 바이올로지(Genome Biology)'에 이날 게재됐다.
환자의 치료과정에서 유전자검사나 약물반응검사 등을 위해 종양조직을 여러 차례 분석하는 일이 불가피하다.
이 때문에 한 번 채취한 종양세포를 자연적으로 보존하고 충분히 증식시켜 여러 검사의 시료로 쓸 수 있도록 하는 환자유래모델(PDMS·patient-derived models)이 활용된다.
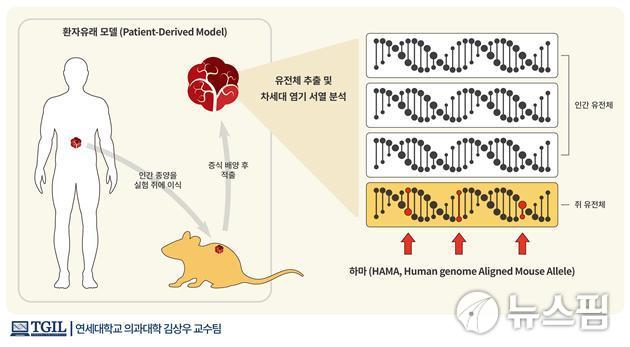
다만, 종양세포를 주로 생쥐(mouse) 체내에서 증식시키거나 생쥐의 세포와 함께 배양하기 때문에 쥐의 세포가 함께 분석돼 자칫 잘못된 결과가 나올 수 있다는 문제점이 있다.
이런 가능성에 대해 꾸준히 제기됐지만 발생 빈도나 예방 방법에 대해서는 알려진 바 없다.
연구진은 환자유래모델에서 있을 수 있는 돌연변이 분석 오류를 찾아내고 나아가 미연에 오류를 방지하는 방법을 개발했다.
우선, 쥐와 사람에게서 나타나는 모든 유전자 서열 차이를 찾고 이를 '하마'(HAMA·human-genome aligned mouse allele) 라고 명명했다.
분석과정에서 이런 '하마'가 나타난다면 질병 관련 유전 변이로 오인할 수 있는데, 생쥐의 유전체 정보로 인한 오류가능성을 한 번 더 확인하도록 안전장치를 제안한 것이다.
특히 잘 알려진 암 관련 돌연변이 데이터베이스의 정보 중 생쥐를 이용한 실험모델에서 비롯된 경우 유독 '하마'의 관찰 빈도가 높게 나타난 것도 확인했다.
나아가 연구진은 유전체 검사 데이터를 통해 나오는 '하마'의 비율을 토대로 환자유래모델에 섞여 있는 쥐 세포의 비율까지 계산할 수 있는 방법을 제시했다.
또한 150가지가 넘는 가상의 오염 데이터를 기반으로 비교 분석을 수행, 최적의 오염 배제 방법을 밝혀냈다.
실제 이를 토대로 최적 유전자분석법을 적용한 결과 기존 분석 대비 정확성을 약 58% 가량 높일 수 있었다는 설명이다.
김상우 교수는 "이번 연구는 체외에서 보존, 증식된 환자 암세포 시료(Specimen)의 유전체 분석과정에서 발생할 수 있는 오류를 바로잡아 향후 더욱 정확한 정보에 기초해 환자를 치료할 수 있는 실마리가 될 것"이라고 말했다.
kimys@newspim.com